Modificaciones a los RNA transcritos
Los distintos RNA transcritos en los eucariontes presentan una serie de especializaciones no encontradas en procariontes. A medida que transcurre la transcripción, las moléculas de mRNA, llamadas transcritos primarios, son modificadas. Esto ocurre antes de que sean transportadas al citoplasma, que es el sitio donde ocurre la traducción. Las modificaciones son varias e incluyen:
1. Adición del CAP
Un nucleótido modificado (CAP) se añade al extremo 5’ del mensajero. Este “casquete” es imprescindible para la unión del mRNA al ribosoma y protege al mRNA de la degradación.
2. Poliadenilación
En el extremo 3’ del mRNA hay una secuencia señal (AAUAAA) a la que se unen factores específicos y la enzima poli-A polimerasa. Esta enzima estimula la escisión en un sitio ubicado 10 a 35 nucleótidos hacia el extremo 3’ de la señal. Luego, la enzima agrega, de a uno, una cola de ribonucleótidos de adenina (cola de poli-A) y así se genera el extremo 3’ del mRNA maduro. Esta cola de poli-A, contiene 200-250 nucleótidos y parecería que influyen en la estabilidad y en la capacidad de que los mRNA sean traducidos en el citoplasma.
3. Corte y empalme o splicing
Durante la transcripción, el mRNA sufre un proceso de corte y eliminación de secuencias que no llevan información llamadas intrones, y el posterior empalme de los exones; secuencias que si llevan información, los. En un primer paso se unen al mRNA inmaduro unas pequeñas partículas de RNA nucleares asociadas con proteínas denominadas snRNP (del inglés, small nuclear ribonucleo-protein particles). Las snRNP se unen a secuencias cortas ubicadas entre los intrones y los exones. Luego se añaden más proteínas y forman un gran complejo con el RNA que se denomina spliceosoma. Además de desempeñar funciones de reconocimiento de esas secuencias, las snRNP llevan a cabo funciones catalíticas.
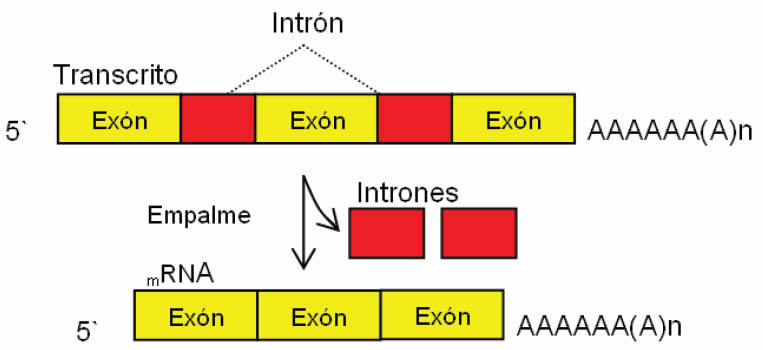
El proceso de splicing, catalizado por spliceosomas, ocurre sólo en organismos eucariotas. Las secuencias que se eliminan son los intrones, mientras que las secuencias que permanecen y forman parte del mRNA maduro son los exones.
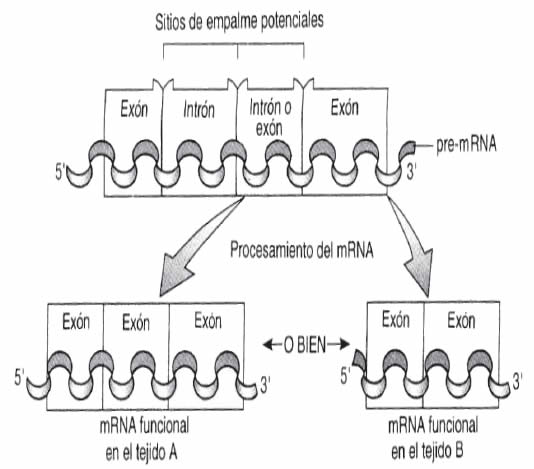
En muchos casos, un mismo transcrito primario o pre mRNA (RNA heteronuclear) puede ser procesado por splicing en más de una forma. Este empalme alternativo permite obtener moléculas de mRNA maduro diferentes a partir de moléculas de mRNA inmaduro originalmente idénticas, lo cual da por resultado polipéptidos con distintas funciones; un mismo gen puede producir una proteína en un tejido y otra distinta en otro tejido.
Esto es posible porque algunos genes producen moléculas de pre-mRNA que tienen múltiples patrones de empalme. Se ha observado que estos pre-mRNA presentan un segmento que puede ser Intrón o Exón. Este procesamiento diferencial del RNA nuclear permite, a las células de cada tejido, producir su propia versión de mRNA correspondiente al gen específico (Figura 9).
El investigador Thonas R. Cech y sus colaboradores, en los Estados Unidos, estudiando el splicing del rRNA en un ciliado de agua dulce llamado Tetrahymena, encontraron que en estos organismos unicelulares eucariontes el propio intrón del rRNA inmaduro actúa como catalizador de la escisión y el empalme, es decir, se produce un empalme autocatalítico. Esta secuencia de RNA se pliega formando una estructura compleja que funciona como enzima, que se ha denominado ribozima.Se han encontrado otros ejemplos de empalme autocatalítico en varios organismos, en RNA codificados por genes mitocondriales o de cloroplastos, en algunos genes nucleares de eucariontes unicelulares y en algunos genes de bacteriófagos, pero no en eucariontes multicelulares.
De gran importancia es el hecho que los genes eucariontes poseen en su estructura exones e intrones, situación no observada en procariontes. El número de intrones varía para cada gen, sin embargo, su número aumenta a medida que el organismo es más complejo y de reciente evolución. Se ha propuesto que los intrones promueven la recombinación genética (vía crossingover), y por lo tanto aumentan la velocidad de evolución (de cambio).